【理論/Python実装】Graphical LASSO【遺伝子発現制御】
目次
- はじめに
- 1. 多変量正規分布と条件付き独立性
- 2. 尤度関数の導出
- 3. Graphical LASSOの最適化問題の導出
- 4. 式変形の詳細解説
- 5. Graphical LASSOの解法
- 6. Graphical LASSOの意義と応用
- 7. Graphical LASSOのPythonによる実装
- 8. ハイパーパラメータの選び方
- 9. 遺伝子発現ネットワークの構造推定の例
- 10. まとめ
- 参考文献
はじめに
**Graphical LASSO(グラフィカルラッソ)**は、多変量正規分布に基づく変数間の条件付き独立性を推定し、スパースなネットワーク構造を復元するための強力な手法です。
特に、高次元データ(変数の数がサンプル数を大きく超えるデータ)において、精度行列(分散共分散行列の逆行列)のスパース推定が重要な課題となります。
本記事では、Graphical LASSOの理論を数式を交えて丁寧に解説し、Pythonによる実装と遺伝子発現制御ネットワークへの応用にも触れます。
1. 多変量正規分布と条件付き独立性
まず、Graphical LASSOが対象とするのは、次元の多変量正規分布: (X \sim \mathcal{N}_p(0, \Sigma))です。
ここで、
-
は正定値の共分散行列
-
は精度行列(逆共分散行列)
です。
多変量正規分布における重要な性質として:
が成り立ちます。つまり、精度行列の非ゼロパターンが、変数間の条件付き独立関係を表すのです。
2. 尤度関数の導出
次に、データ (サンプル、次元)の対数尤度関数を導出します。
多変量正規分布の確率密度関数は
です。
これを 個の独立なサンプル に対して対数を取ると
となります。
定数項を除いてまとめると
ここで、 は標本共分散行列:
です。
3. Graphical LASSOの最適化問題の導出
3.1 最大対数尤度推定(MLE)
LASSOの前に、通常の最大対数尤度推定(MLE)の問題は次の通りです
\max\_{\Theta \succ 0} \ \left{ \log |\Theta| - tr(S \Theta) \right}これは共分散行列の逆行列を直接推定する問題です。
しかし、高次元()の場合、は特異行列であり、直接の推定は不可能です。
3.2 L1正則化の導入(Graphical LASSO)
ここで、スパース性を導入するために、L1正則化を加えます。以下がGraphical LASSOの目的関数です。
\max\_{\Theta \succ 0} \ \left{ \log |\Theta| - tr(S \Theta) - \lambda \sum\_{i \neq j} |\theta\_{ij}| \right}または最小化形式に書き換えると
\min\_{\Theta \succ 0} \ \left{ -\log |\Theta| + tr(S \Theta) + \lambda |\Theta|\_1 \right}となります。
ここでは精度行列のオフダイアゴナル要素のL1ノルムです。
4. 式変形の詳細解説
4.1 ログ行列式の性質
まず、行列式の対数は以下のような性質を持ちます。
ここで、 は の固有値です。
精度行列は正定値なので、固有値は全て正です。
4.2 トレースの性質
です。トレースは行列積の要素積の和であり、対称性のある二次形式において重要な役割を果たします。
4.3 正則化項の意味
L1ノルム正則化項は、精度行列の非対角成分(条件付き依存関係を示す部分)のスパース化を促します。
特に、を大きくすると、多くの要素がゼロ化されます。
4.4 Graphical LASSOの目的関数まとめ
Graphical LASSOの最終的な目的関数は次のようになります。
この問題は、**対数尤度の最大化(モデルのあてはまりの良さ)**と、**L1ノルムによるスパース性の強制(過学習の防止)**のトレードオフを解く問題です。
5. Graphical LASSOの解法
この最適化問題は凸最適化問題であり、Friedmanら(2008)の論文で提案された「ブロック座標降下法」によって解かれます。基本的なアイデアは次の通りです:
-
Θ\Theta の1行1列を固定し、他の行列成分を条件付きで最適化
-
サブ問題はLASSO回帰に帰着し、既存のアルゴリズムで解ける
-
各変数について反復的に更新
詳細な解法アルゴリズムは専門的な内容になるため、別記事で解説します。
6. Graphical LASSOの意義と応用
Graphical LASSOは、以下のような問題に応用されています:
-
遺伝子ネットワークの推定
-
脳神経活動ネットワークの解析(fMRIデータ)
-
金融市場におけるリスクネットワークの構造推定
-
気象データやIoTセンサーデータの依存構造解析
特に、**高次元小サンプル問題()**での安定したネットワーク推定が可能な点が強力です。
7. Graphical LASSOのPythonによる実装
Pythonでは、sklearnやskggm、QUICライブラリなどを用いてGraphical LASSOを実装できます。ここでは、sklearn.covarianceモジュールのGraphicalLassoクラスを使った例を紹介します。
7.1 必要なライブラリのインポート
import numpy as npimport matplotlib.pyplot as pltfrom sklearn.covariance import GraphicalLassoimport seaborn as sns7.2 データの生成
まずは、ダミーデータを生成します。
np.random.seed(0)n_samples, n_features = 100, 5
# 相関構造を持つ共分散行列prec = np.array([ [1.0, -0.4, 0, 0, 0], [-0.4, 1.0, -0.3, 0, 0], [0, -0.3, 1.0, -0.2, 0], [0, 0, -0.2, 1.0, -0.1], [0, 0, 0, -0.1, 1.0]])
cov = np.linalg.inv(prec)X = np.random.multivariate_normal(mean=np.zeros(n_features), cov=cov, size=n_samples)Xの形状と中身は以下です。
X.shape# (100, 5)
X[:, :5]# array([[-1.51825267, -2.28955998, 0.64205379, -2.47082997, -0.26380282],# [ 1.43948198, 0.91083282, 0.25170104, -0.44917451, -0.23546759],# [ 0.45744412, -0.06202684, -0.25803166, -1.27957377, -1.19800932],# [ 0.00979201, 0.51196984, -1.23243424, -1.2820214 , -0.37795672],# [ 3.21682083, 1.25318879, 2.34005276, 0.31735484, -1.05566578]])7.3 Graphical LASSOの適用
model = GraphicalLasso(alpha=0.2, max_iter=100)model.fit(X)
# 推定された精度行列Theta_est = model.precision_7.4 結果の可視化
推定された精度行列のヒートマップを表示します。
plt.figure(figsize=(6, 5))sns.heatmap(Theta_est, annot=True, fmt=".2f", cmap="coolwarm", square=True)plt.title("Estimated Precision Matrix (Graphical LASSO)")plt.show()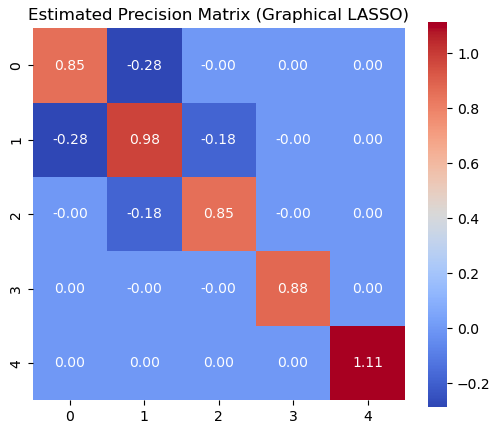
8. ハイパーパラメータの選び方
Graphical LASSOにおける重要なパラメータは正則化パラメータ です。
この値を大きくするとスパース性が強まり、より多くの要素がゼロになります。
一方で、値を小さくするとより密な推定結果になります。
のチューニングには以下のような方法があります。
-
**交差検証(Cross-Validation)**を用いる
-
BICやAICを基準にモデル選択
-
ドメイン知識に基づく設定
例: sklearn.covariance.GraphicalLassoCV クラスを用いると自動でのチューニングが可能です。
from sklearn.covariance import GraphicalLassoCV
model_cv = GraphicalLassoCV()model_cv.fit(X)print("Optimal alpha:", model_cv.alpha_)9. 遺伝子発現ネットワークの構造推定の例
以下は、ある転写因子のmRNA量をその他の転写因子の線形結合でモデル化しLASSO等を用いて係数を決め、各転写因子をノードに係数をエッジの重みとしたグラフになります。
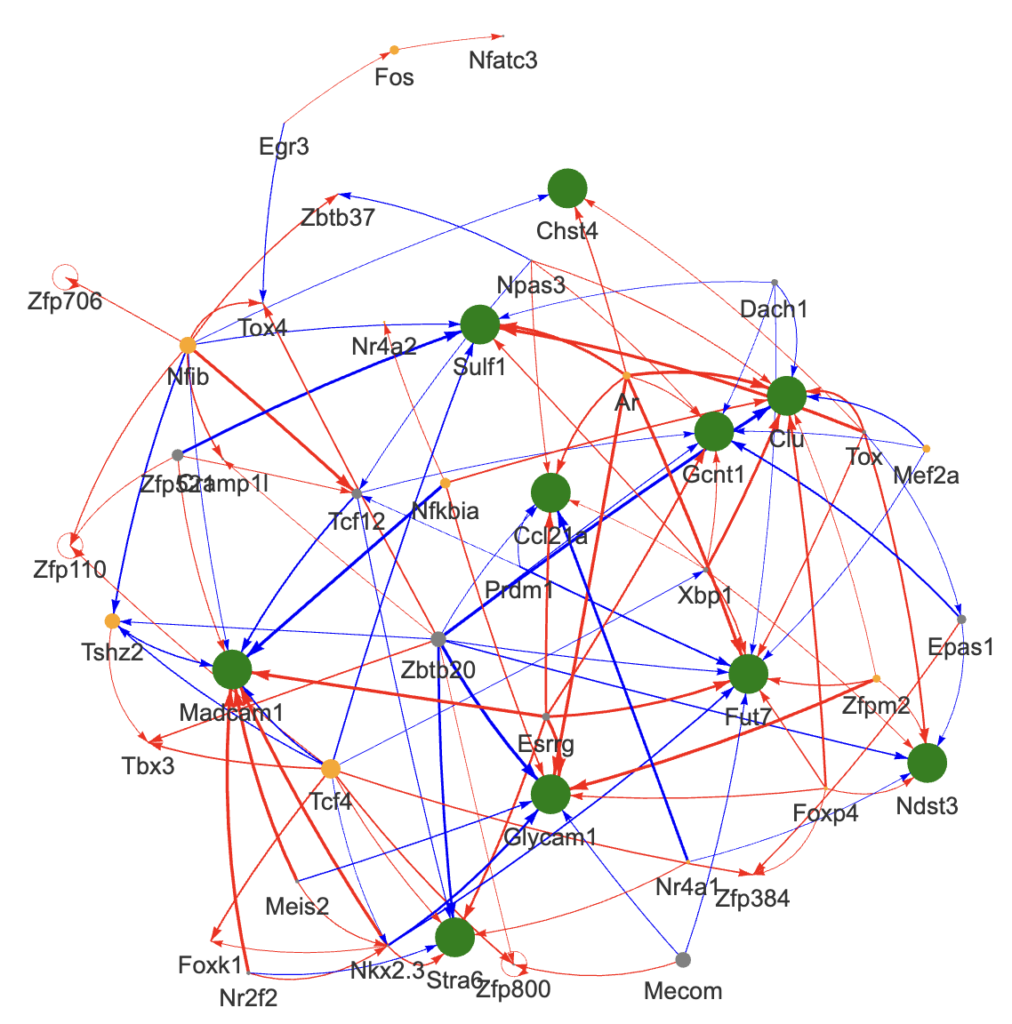
当然この方法では、間接的な制御が直接的な制御としても現れます。
例えば、A → B → Cという制御であった場合、A → B → Cというエッジに加え、A → Cというエッジも現れます。
この間接的なみかけの制御を排除するためにGraphical LASSOを利用してみます。
9.1. 入力データ
列に転写因子、行に細胞が対応する二次元データを用意します(列目の行目は転写因子の番目の細胞におけるmRNA量となるようなテーブルデータ)。
import pandas as pd
# 以下は例df_X = pd.DataFrame([[1, 2, 3], [4, 5, 6], [7, 8, 9]])df_X.columns = ['Ar', 'Esrrg', 'Nr2f2']df_X.index = ['cell1', 'cell2', 'cell3']df_X| Ar | Esrrg | Nr2f2 | |
|---|---|---|---|
| cell1 | 1 | 2 | 3 |
| cell2 | 4 | 5 | 6 |
| cell3 | 7 | 8 | 9 |
9.2. Graphical LASSOの実施と相関行列の作成
# np.arrayに変換X = np.array(df_X)
# GraphicalLassoCV を実行する。model = GraphicalLassoCV(alphas=4, cv=5)model.fit(X)
# 分散共分散行列を取得する。# -> 参考URL:https://scikit-learn.org/stable/modules/generated/sklearn.covariance.GraphicalLassoCV.htmlcovariance_matrix = model.covariance_
# 分散共分散行列を相関行列に変換する。diagonal = np.sqrt(covariance_matrix.diagonal())correlation_matrix = ((covariance_matrix.T / diagonal).T) / diagonal9.3. 見かけのエッジリストを作成
# グラフ表示のために対角成分が0の行列を生成する。correlation_matrix_diag_zero = correlation_matrix - np.diag(np.diag(correlation_matrix) )df_grahp_data = pd.DataFrame(index=genes, columns=genes, data=correlation_matrix_diag_zero.tolist())
# negative listを作成negative_list = []for i in range(len(df_grahp_data)): for j in range(i + 1, len(df_grahp_data)): if abs(df_grahp_data.iloc[i, j]) < cutoff: negative_list.append([genes[i], genes[j]])9.4. 見かけのエッジを除去したグラフの描画
# 1. フィルター前のエッジに関する二次元配列のデータ# 各行が1つのエッジに対応。1列目はtargetのnode名、2列目はsourceのnode名、3列目はedgeの重さ、4列目はエッジの色# 今回の場合、転写因子Aに対しA以外の転写因子の線形結合をXのデータを用いてLASSOでモデル化した際に係数が0でなければAがtargetで係数が0でなかった転写因子がsource# なおその際の係数がedgeの重さで色は係数の符号に対応。# 以下は「エッジに関する二次元配列のデータ」の例。本来は上述のようにしてXからLASSOなどで決める。edge_list = [['Esrrg', 'Ar', 3, 'red'], ['Nr2f2', 'Ar', 2, 'blue']] # , ...]
# 2. negative_listのエッジを除くprint(f'Graphical Lasso: {len(edge_list)} --> {len(edge_list) - len(negative_list)}')filtered_edge_list = []for edge in edge_list: # filter by graphical lasso target = edge[0] source = edge[1] if target != source: if [target, source] in gl_neg_list or [source, target] in gl_neg_list: continue # add edges filtered_edge_list.append(edge)
# 3. ノードのリストを作成nodes_list = []for e in filtered_edge_list: nodes_list.extend([e[0], e[1]])nodes_list = list(set(nodes_list))
# 4. dataframe化# edgesdf_edges = pd.DataFrame(filtered_edge_list, columns=["target", "source", "weight", "color"])# nodesdf_nodes = pd.DataFrame({ 'node': nodes_list, 'color': [ # blue_list, red_list, green_list(ノードの色)は各々で好きなように定義 'blue' if g in blue_list else 'red' if g in red_list else 'green' if g in green_list else 'gray' for g in nodes_list ], 'size': 20 })
# 5. グラフを描画 & 保存# pyvisでグラフを描画import networkx as nxfrom pyvis.network import Network# df_edgesからグラフ作成G = nx.from_pandas_edgelist(df_edges, source="source", target="target", edge_attr=True, create_using=nx.DiGraph())# ノードの情報を加えるnode_attr_dic = df_nodes.set_index("node").to_dict(orient="index")nx.set_node_attributes(G, node_attr_dic)# グラフ描画pyvis_G = Network(notebook=True)pyvis_G.from_nx(G)pyvis_G.show_buttons(True)pyvis_G.show(f'filtered_network.html')見かけのエッジを除いたところ、以下のようなすっきりしたグラフになりました。
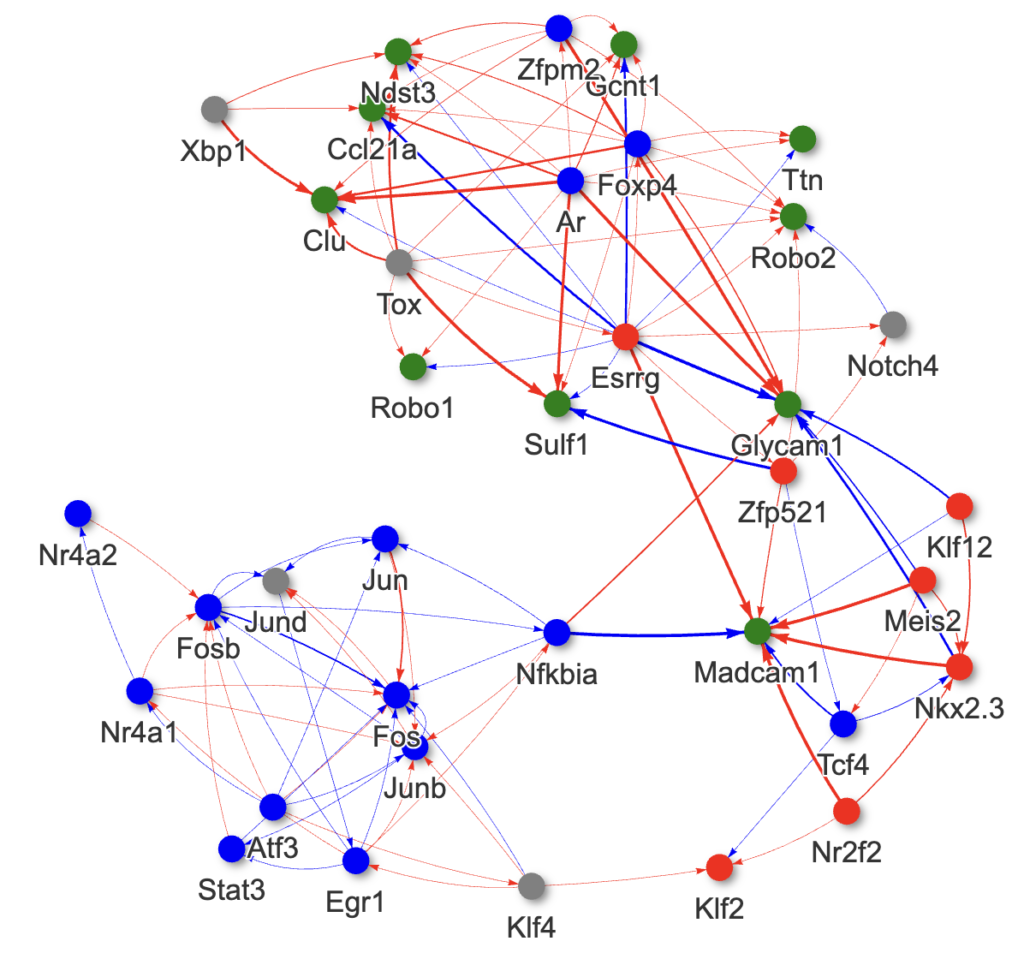
10. まとめ
Graphical LASSOは、高次元データにおける変数間の条件付き独立性を推定し、スパースなネットワーク構造を明らかにする強力な手法です。
Pythonを用いれば、sklearnライブラリで手軽に実装できるため、応用範囲は広がります。
参考文献
-
Friedman, J., Hastie, T., & Tibshirani, R. (2008). Sparse inverse covariance estimation with the graphical lasso. Biostatistics, 9(3), 432–441.
-
Witten, D. M., Friedman, J. H., & Simon, N. (2011). Statistical learning techniques for high-dimensional data. Springer.
-
Scikit-learn documentation: GraphicalLasso

