
In-Fusionクローニング
目次
原理
In-Fusionクローニングのメカニズムは、リコンビナーゼがDNA断片とベクターの間の特定の15bp同一配列を認識し、結合することにより機能します。この際、結合は非常に高精度で行われ、遺伝子操作の誤りをほぼ排除します。
特徴
使用する任意のベクターの末端配列を利用してクローニングを行うため、あらゆるベクターが使用でき、余分な配列が一切付加されず、しかもディレクショナルクローニングを行うことができる優れた手法です。
実験手順
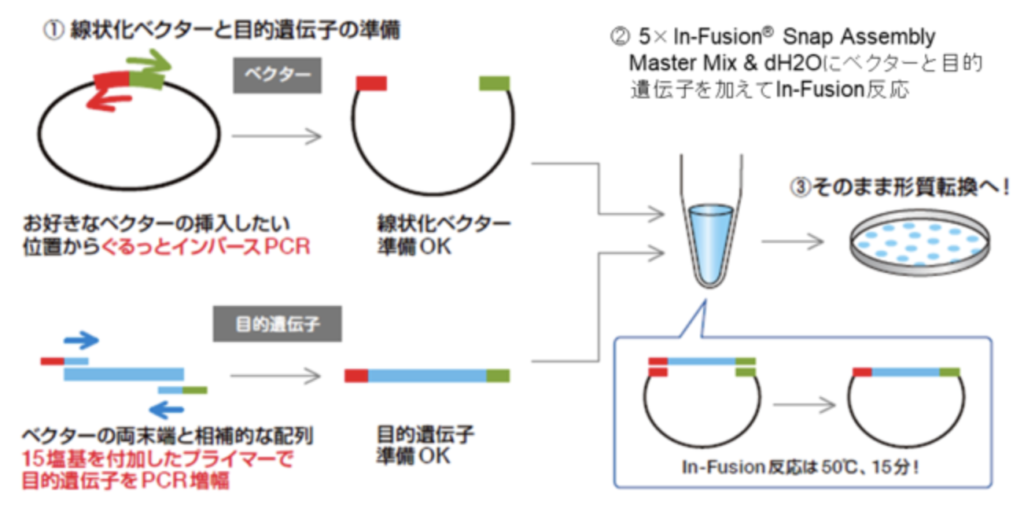
In-Fusionクローニングの手順は以下の通りです:
-
- PCR増幅を行って目的のDNA断片を取得します。このとき、PCRプライマーの設計を工夫して、DNA断片の両端にはIn-Fusionクローニングに必要な15bpの配列を追加します。
-
- 使用したいベクターに対して、上述の15bpを追加する形でインバースPCRを行います。
-
- 次に、リコンビナーゼを含むIn-Fusionミキシングを用いて、DNA断片とベクターDNAを混合します。
-
- 特定の条件下でのインキュベーションにより、リコンビナーゼが活性化し、DNA断片とベクターDNAの間の結合が始まります。
-
- 結合が完了すると、In-Fusionミキシングを停止し、DNA断片がベクターに結合した生成物が得られます。
TAクローニングとの比較
In-Fusionクロニングは速度や精度の点で、従来の遺伝子クローニング手法、例えば、リガーゼ依存的な方法やTAクローニングなどと比較して優れています。
ただし、In-Fusionクローニングキットは他の遺伝子クローニング手法と比較して少々高価です。
Gibson Assemblyとの比較
Gibson Assemblyもまた、複数の遺伝子断片を効率的に接合するクローニング手法です。
両方は多くの共通点を持っていますが、主な違いはGibsonアッセンブリはプロトコルがやや複雑であり、結果の精度が少し劣る点です。
しかし、Gibsonアッセンブリではより長いオーバーラップシーケンス(通常は20-40bp)を使用するため、より大きな二つ以上のフラグメントを接合するのに適しています。

